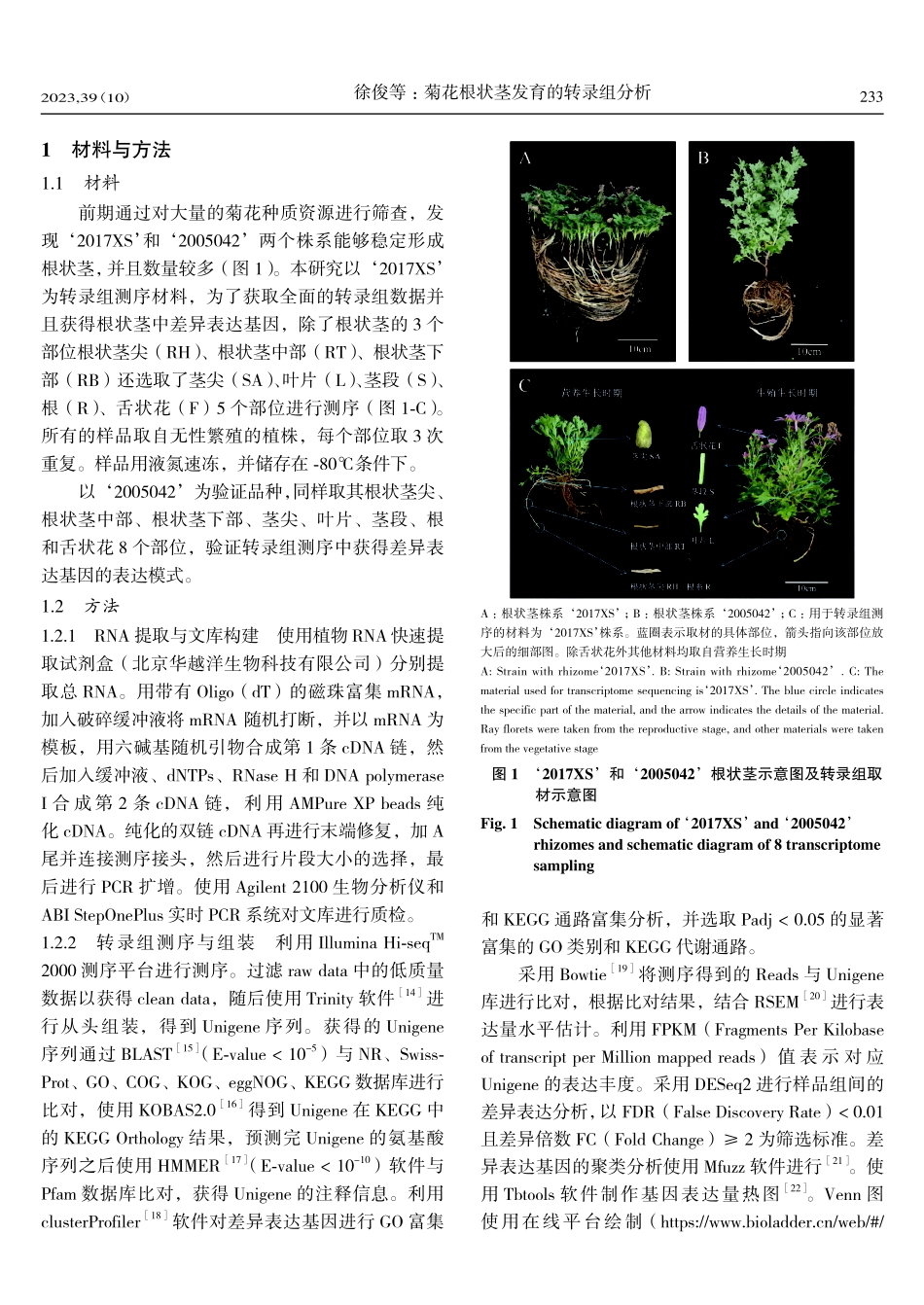
·研究报告·生物技术通报BIOTECHNOLOGYBULLETIN2023,39(10):231-245收稿日期:2023‑04‑08基金项目:北京市植物园管理处科技课题(BZ202203)作者简介:徐俊,男,硕士研究生,研究方向:花卉种质资源与遗传育种;E‑mail:1405549321@qq.com通讯作者:黄河,男,博士,教授,研究方向:花卉分子生物学;E‑mail:101navy@163.com;张蒙蒙,女,博士,高级工程师,研究方向:菊属植物种质资源创新与育种;E‑mail:mengmengzhang818@sina.com菊花根状茎发育的转录组分析徐俊1叶雨晴1牛雅静2黄河1张蒙蒙2(1.北京林业大学园林学院花卉种质资源创新与分子育种北京市重点实验室国家花卉工程技术研究中心城乡生态环境北京实验室,北京100083;2.北京市植物园管理处北京市花卉园艺工程技术研究中心,北京100093)摘要:为了探究菊花根状茎形成的分子机制,本研究选取了具有稳定根状茎的菊花株系‘2017XS’的根状茎尖、根状茎中部、根状茎下部、叶片、茎段、根系、茎尖、舌状花8个部位进行了转录组测序,并利用生物信息学方法对根状茎尖和整个根状茎中高表达的基因进行筛选。转录组测序共得到159.51GB数据,组装后得到100235个Unigene。其中,64956(64.80%)个Unigene在7个公共蛋白质数据库中得到了注释。为了找到根状茎发育的关键基因,通过加权基因共表达网络分析(weightedcorrelationnetworkanalysis,WGCNA)、K‑means和基于Venn筛选差异基因3种方式筛选在根状茎尖和根状茎中高表达基因。最终筛选到20个在根状茎尖中高表达的基因和36个在根状茎中高表达的基因。这些基因包括了和植物非生物胁迫相关的基因、脱落酸(abscisicacid,ABA)代谢基因、红光受体和紫外光受体以及光周期核心转录因子,选取6个差异表达较显著的基因进行RT‑qPCR荧光定量分析,结果与转录组测序数据一致,验证了转录组的有效性。且这些差异表达基因在同样具根茎的菊花株系‘2005042’中亦表现为在根状茎中特异高表达。综上,菊花根状茎的形成和发育可能受到包括脱落酸在内的植物激素以及光周期等因素的影响,本研究对进一步探究菊花根状茎发育的分子机制提供了重要依据。关键词:根状茎;转录组;内源激素;光周期DOI:10.13560/j.cnki.biotech.bull.1985.2023‑0315TranscriptomeAnalysisofRhizomeDevelopmentinChrysanthemum××morifoliumXUJun1YEYu‑qing1NIUYa‑jing2HUANGHe1ZHANGMeng‑meng2(1.SchoolofLandscapeArchitecture,BeijingForestryUniversity,BeijingKeyLab...